

DNA与蛋白质的相互作用对于DNA复制、染色质形成、转录调控等许多关键细胞进程至关重要,这些细胞过程需要DNA展现出不同程度的柔性来缠绕蛋白质,其中起关键作用的是100-bp以下的DNA片段的柔性 (1)。实验上,可以通过loop-seq等方法获取DNA片段的柔性。但是,使用这些方法在全基因组尺度上低成本地获取DNA序列的柔性仍然具有一定的困难 (2,3)。
2023年9月11日,美高梅登录正网精准医疗多组学研究中心/美高梅登录正网肿瘤医院吴华君课题组,上海交通大学公共卫生学院单细胞组学与疾病研究中心郑小琪课题组以及北京协和医院疑难、重症与罕见病全国重点实验室黄超兰课题组合作在Nucleic Acids Research(IF=14.9)上发表了题为“Assessing base-resolution DNA mechanics on the genome scale”的研究论文,利用AI技术实现对任意DNA序列柔性的快速低成本预测,为研究DNA力学性质参与转录调控的机制提供了有力工具(图1)。
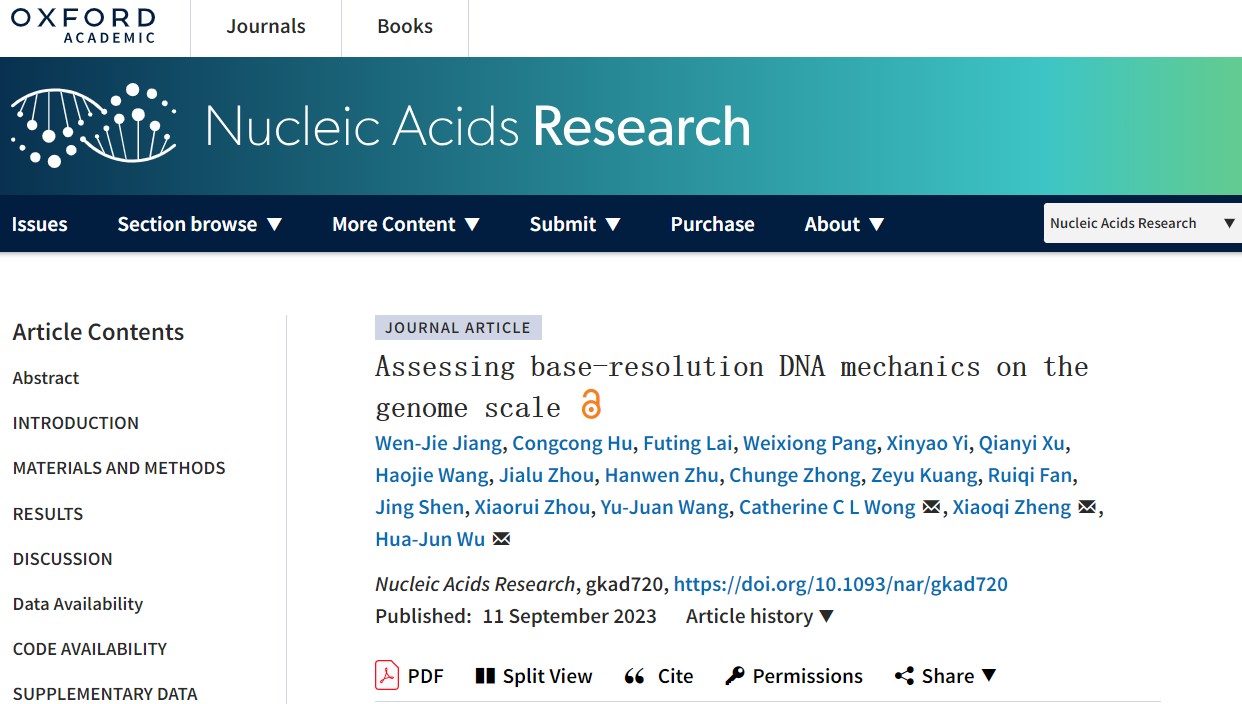
图1.文章摘要图
文中提出了一种基于深度学习的方法——BendNet,通过使用无路由的多胶囊网络 (4) 提取编码DNA柔性的序列特征,每条50-bp的DNA序列经过独热编码,通过三种不同深度的卷积层提取序列的不同高维特征,再由三个胶囊全连接网络来预测DNA序列的柔性(图2)。
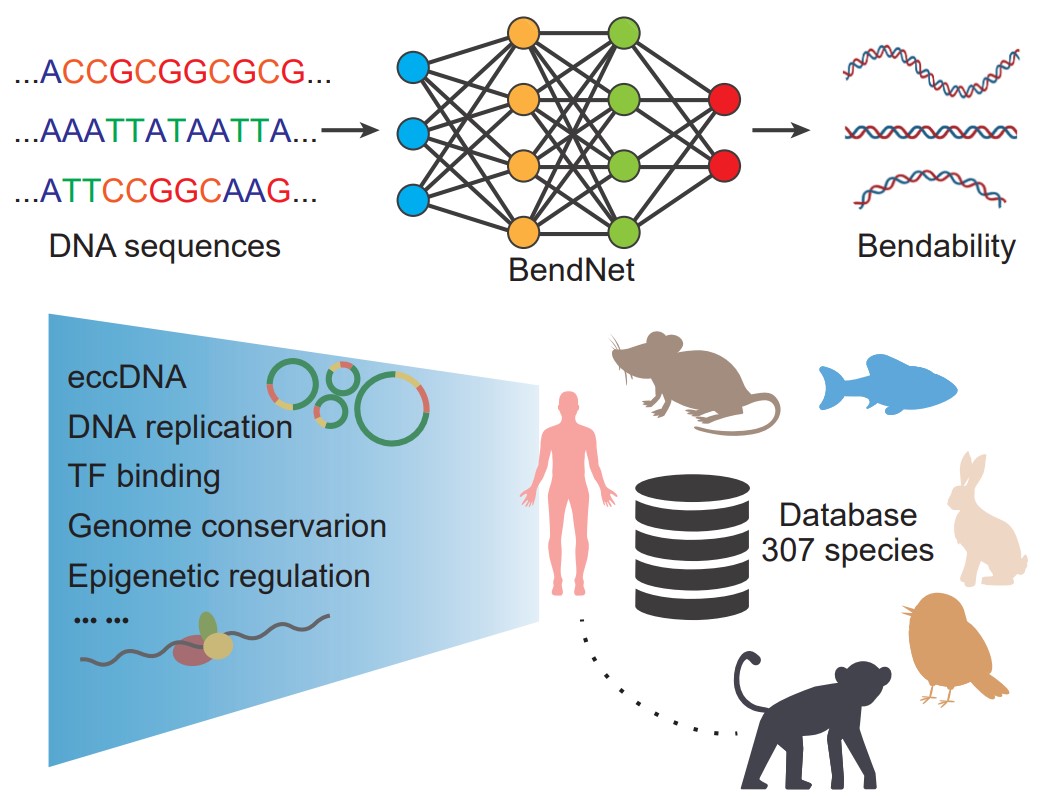
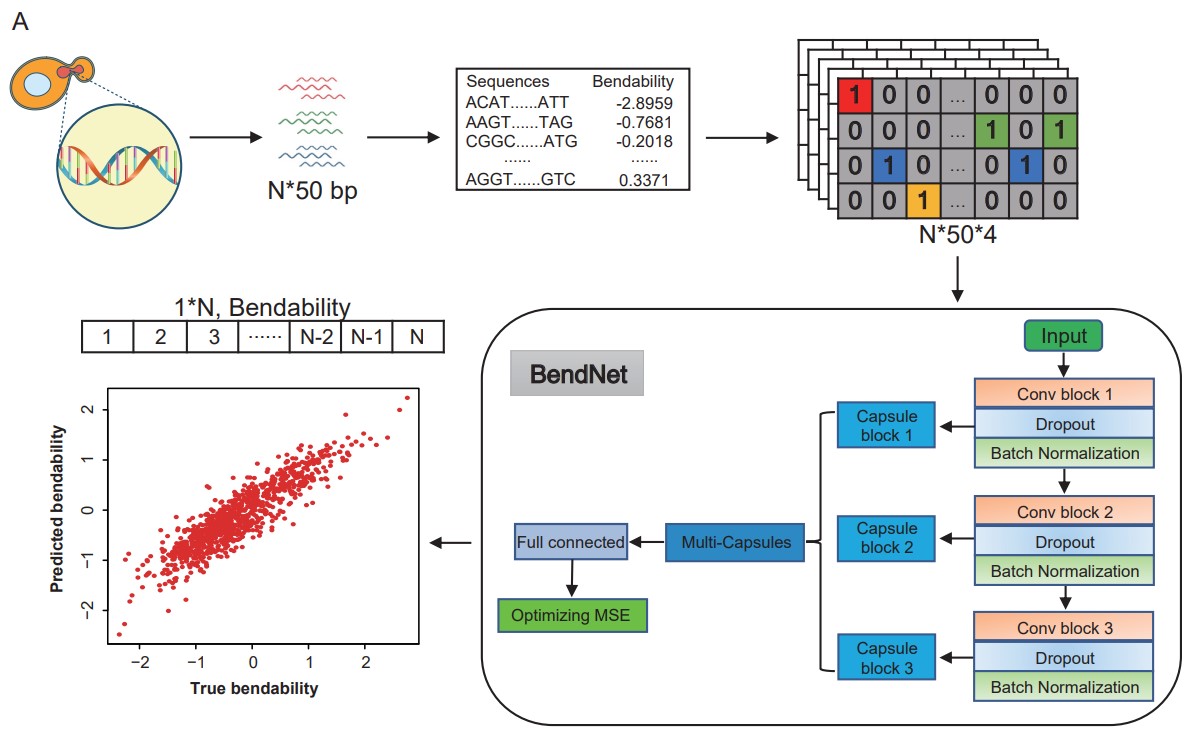
图2.BendNet架构概念图
作者将BendNet与经典的机器学习和深度学习模型进行了比较,包括Random Forest(RF), Support Vector Regression(SVR), AlexNet, ResNet, GoogleNet, VGG, DNAcycP and DeepBend。BendNet不仅在预测准确性方面表现出色,并且,它在训练和预测过程中的速度均领先其他深度学习模型,比第二快的模型节省了38%的训练时间和25%的预测时间。BendNet也表现出了很好的泛化能力,它在不同实验类型、不同物种的数据集上表现优异。这些结果表明BendNet是一个准确、高效且具有广泛适用性的模型,未来随着实验数据的积累可以应用于预测其他DNA性质,例如DNA扭曲、超螺旋和扭转刚度等,对于DNA特性研究具有广泛的应用价值。
随后,作者将BendNet应用于人类基因组,首次获得了全基因组尺度、单碱基分辨率的DNA 柔性图谱。研究发现,DNA序列在转录起始/终止位点等区域的柔性较低、在增强子区域附近偏高。在活跃的转录调控区域中GC-Content与DNA柔性均呈正相关。DNA柔性区域还富集疾病相关的风险位点。此外,绝大部分转录因子(TF)结合于刚性(柔性低)的DNA序列上,但某些先锋转录因子和与染色质结构相关的转录因子,如EBF1和CTCF,在其结合区间中呈现出异常高的柔性。这些转录因子与核小体共同结合或竞争结合,以发挥其功能。尤其CTCF结合区域的DNA的柔性明显高于其它区域,这可能暗示这些区域在捕获并维持CTCF在特定位置形成DNA拓扑结构域(Topologically associating domain)的作用上具有重要意义。
该研究为大规模DNA序列柔性的计算提供了有力的工具,并且加深了DNA力学性质在染色质调控中作用的理解。为构建一个更为全面的DNA 柔性资源库,作者利用BendNet预测了307个物种的基因组,将其存储于Zenodo服务器上,并为科研人员开发了一个用户友好的BendNet使用网站(http://www.dnabendnet.com/)(图3)。
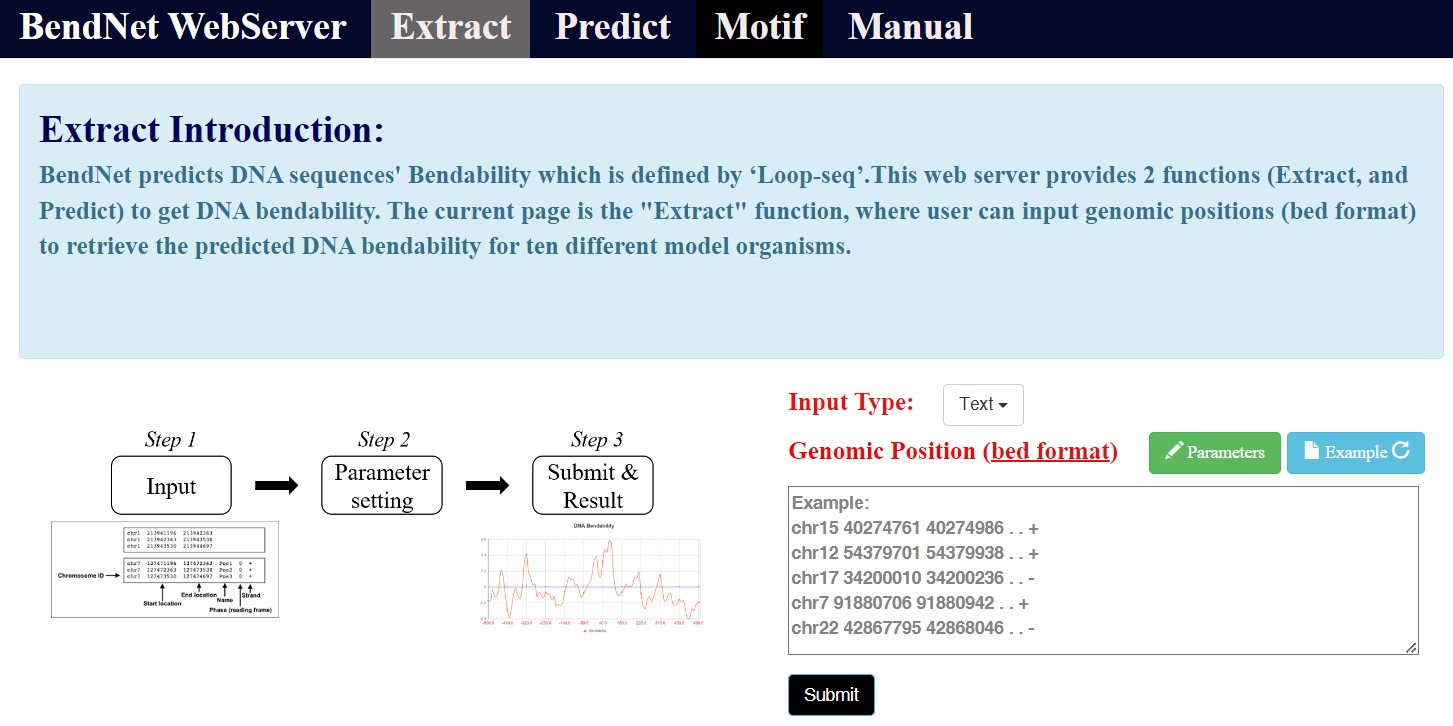
图3.BendNet网站首页
美高梅登录正网肿瘤医院蒋文杰为论文的第一作者。北京协和医院疑难、重症与罕见病全国重点实验室黄超兰教授,上海交通大学公共卫生学院单细胞组学与疾病研究中心郑小琪教授,和美高梅登录正网精准医疗多组学研究中心/美高梅登录正网肿瘤医院吴华君研究员为该论文的共同通讯作者。该研究得到了国家自然科学基金、上海市自然科学基金和国家重点研发计划项目的资助。
原文链接:https://academic.oup.com/nar/advance-article/doi/10.1093/nar/gkad720/7269185?searchresult=1
Reference
1. Vafabakhsh, R. and Ha, T. (2012) Extreme bendability of DNA less than 100 base pairs long revealed by single-molecule cyclization. Science, 337, 1097-1101.
2. Basu, A., Bobrovnikov, D.G., Qureshi, Z., Kayikcioglu, T., Ngo, T., Ranjan, A., Eustermann, S., Cieza, B., Morgan, M.T. and Hejna, M. (2021) Measuring DNA mechanics on the genome scale. Nature, 589, 462-467.
3. Tang, L. (2021) Sequencing DNA bendability. Nature Methods, 18, 121-121.
4. Sabour, S., Frosst, N. and Hinton, G.E. (2017), Proceedings of the 31st International Conference on Neural Information Processing Systems. Curran Associates Inc., Long Beach, California, USA, pp. 3859–3869.
(美高梅登录正网肿瘤医院)