

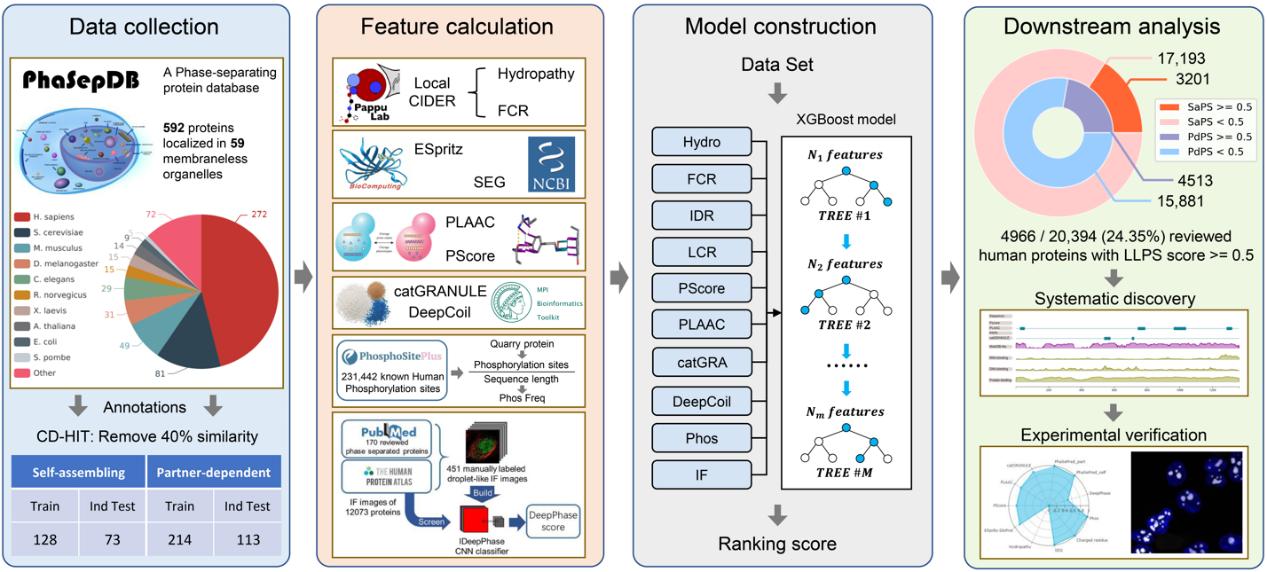
近日,美高梅登录正网基础医学院医学生物信息学系李婷婷研究组与清华大学生命科学学院李丕龙研究组在PNAS杂志上在线发表文章Screening Membraneless Organelle Participants with Machine Learning Models that Integrate Multimodal Features,开发了相分离蛋白预测工具PhaSePred(http://predict.phasep.pro/)。

相分离(phase separation, PS)是调控生物大分子(如蛋白质,核酸)在细胞中区室化分布的重要机制之一。根据参与相分离过程的不同机制可以将相分离蛋白划分为两类:一类能够自发组装形成相分离凝聚体(self-assembly);另一类则依赖与其他生物大分子的相互作用发生相分离(partner-dependent)。目前已有相分离蛋白预测工具能较好预测自组装相分离蛋白(PS-Self),但在预测互作依赖相分离蛋白(PS-Part)时表现欠佳。因此,有必要开发一种新预测工具来筛选潜在的互作依赖相分离蛋白。
鉴于此,课题组基于此前发表的相分离蛋白数据库PhaSepDB,将相分离蛋白分为自发相分离蛋白(hSaPS)与互作依赖相分离蛋白两类集两类(hPdPS),并在多模态特征上比较发现了两个相分离蛋白集合和背景蛋白集合之间的分布存在显著差异。在整合多模态特征分别构建自组装相分离蛋白预测工具SaPS和互作依赖相分离蛋白预测工具PdPS后,课题组在人类蛋白独立测试集上进行模型评估,结果表明结合了多模态特征的模型与现有方法相比具有更加优异的性能,例如与代表性相分离预测工具catGRANULE、PLAAC和PScore相比,PdPS模型在预测互作依赖相分离蛋白的表现分别提高了12%、20%和20%。
课题组进一步进行了体外相分离实验以测试预测模型的有效性。其中,DHX9被预测可自发发生相分离,实验结果表明DHX9可以在体外自发发生相分离并形成具有一定流动性的液滴。Ki-67被预测可以自发发生,也可以通过相互作用发生相分离,同时Ki-67的已知互作蛋白NIFK被预测可通过相互作用发生相分离,实验结果表明Ki-67在与DNA混合时能在相对较低浓度下发生相分离,而NIFK可以与DNA和Ki-67共同发生相分离。
课题组发现,虽然SaPS和PdPS模型中整合了PLAAC、PScore、catGRANULE等相分离打分工具,但磷酸化修饰、免疫荧光图像等新特征的引入导致上述几个模型所预测的高打分相分离蛋白交集并不多。为此,课题组整合了SaPS、PdPS以及常见相分离蛋白预测工具开发了相分离蛋白预测网站PhaSePred。目前该网站提供了来自18个物种十万多条蛋白的氨基酸级别打分,帮助研究人员快速锁定目标相分离蛋白。
课题组硕士研究生陈钊铭、八年制博士研究生侯超、清华大学生命科学学院王亮博士为本论文共同第一作者。
李婷婷课题组致力于通过生物信息学手段研究相分离过程,目前已构建相分离蛋白数据库PhaSepDB,相分离/无膜细胞器相关疾病数据库MloDisDB,相分离蛋白预测工具PhaSePred,系列网站(http://lab.phasep.pro/)日均访问量超千次。
原文链接:https://www.pnas.org/doi/10.1073/pnas.2115369119
(美高梅登录正网基础医学院)